DNA-Sequenzierung ist das Verfahren zur Bestimmung der Sequenz von Nukleotidbasen (A, C, T, und G) in einem DNA-Stück. Heutzutage ist es mit der richtigen Ausrüstung und den richtigen Materialien relativ einfach, ein kurzes Stück DNA zu sequenzieren. Durch weitere bioinformatische Auswertung kann eine Artidentifizierung erfolgen. In diesem Text geht es um die Identifizierung von im Boden befindlichem Pilzmyzel möglichst bis zur Pilzart durch DNA-Sequenzierung und angeschlossener bioinformatischer Analyse mittel OTU Clustering und Datenbankabgleich via BLAST.
Wir haben die Analyse eines kompletten Bodenprofils, das mit einem Bohrstock entnommen wurde, durchführen lassen. Ziel der Untersuchung war es, Informationen über die vertikale, artspezifische Verteilung von Pilzmyzelien in dem Waldboden zu erhalten: In welcher Bodentiefe kommt Myzel von der Pilzart X vor. Besonders gespannt waren wir auf die Tiefenverteilung von Hirschtrüffel. Da wir auch die Verteilung von Cäsium 137 im Boden detailliert in dem Bodenprofil ermittelt haben, sollten mögliche Zusammenhänge mit den Cs 137 Gehalten in Fruchtkörpern von Hirschtrüffel ermittelt werden.
Die Sequenzierung eines gesamten Genoms (der gesamten DNA eines Organismus) bleibt eine komplexe Aufgabe. Dazu muss die DNA des Genoms in viele kleinere Teile zerlegt, die Teile sequenziert und die Sequenzen zu einem einzigen langen “Konsens” zusammengesetzt werden. Dank neuer Methoden, die in den letzten zwei Jahrzehnten entwickelt wurden, ist die Genomsequenzierung jetzt viel schneller und kostengünstiger möglich als während des Human Genome Project.
Erklärung des Begriffs OTU (Operational Taxonomic Unit)
Ein gängiger Ansatz zur Erforschung der genetischen Vielfalt in einer Pilz Gemeinschaft besteht darin, die 16S-rRNA-Sequenzen auf der Grundlage von Sequenzähnlichkeiten in operative taxonomische Einheiten (Operational Taxonomic Units, OTUs) zu gruppieren. Die abgeleiteten OTUs können dann verwendet werden, um Pilzarten, Zusammensetzung von Pilzgemeinschaften und in geringerem Maße auch quantitative Vorkommen abzuschätzen.
Der Begriff: In der traditionellen numerischen Taxonomie wird der Begriff operative taxonomische Einheit (Operational Taxonomic Unit, OTU) verwendet, um eine Anzahl eng verwandter Individuen (z. B. Arten oder Gattungen) zu klassifizieren.
Methoden aus der numerischen Taxonomie werden häufig für Markergen-Sequenzierungsstudien der nächsten Generation angewendet, bei denen Organismen nicht direkt beobachtet werden. Eine OTU wird in der Regel als eine Gruppe von Lesevorgängen mit 97%iger Ähnlichkeit definiert, mit der Erwartung, dass diese ungefähr der Art entsprechen. Dies ist unter der Voraussetzung sinnvoll, dass bei der nachgelagerten Analyse berücksichtigt wird, dass die Korrespondenz von OTUs mit Arten fehlschlagen kann, weil:
- einige Arten haben Gene, die zu mehr als 97% ähnlich sind, was zusammengeführte OTUs ergibt, die mehrere Arten enthalten,
- eine einzelne Art kann zu weniger als 97% ähnliche Paralogs aufweisen, die dazu führen, dass die Art auf zwei oder mehr OTU aufgeteilt wird, und
- Einige Cluster, sogar die Mehrheit, können aufgrund von Artefakten, einschließlich Lesefehlern und Chimären, falsch sein.
Das Basic Local Alignment Search Tool (BLAST )
Das BLAST-Tool (Basic Local Alignment Search Tool) findet Regionen mit lokaler Ähnlichkeit zwischen Sequenzen. Das Programm vergleicht Nukleotid- oder Proteinsequenzen mit Sequenzdatenbanken und berechnet die statistische Signifikanz von Übereinstimmungen. BLAST kann verwendet werden, um funktionelle und evolutionäre Beziehungen zwischen Sequenzen abzuleiten und Mitglieder von Genfamilien zu identifizieren.
RNA-seq- und DNA-seq-Experimente generieren zig Millionen von Lesevorgängen, die aus Transkripten oder Genomen entnommen wurden. Die resultierenden Daten ermöglichen Untersuchungen, die Genexpression, Genstruktur, Nukleotid- und Strukturvariationen umfassen, aber nicht darauf beschränkt sind. Für einige Untersuchungen stehen unterschiedliche Analyseansätze zur Verfügung. Beispielsweise kann die Genexpression mit Alignments oder mit Alignment-freien Methoden untersucht werden.
Artbestimmung von Pilzmyzelien in einem Bodenprofil via DNA-Sequenzierung, OTU-read und BLAST
Methode
DNA Auswertung eines Bodenprofils, Nationalpark Bayerischer Wald, Entnahme September 2018.
Schichtfolgen 2 cm. Humushorizonte ausgebildet. Ah beginnt bei 10 cm Tiefe.
Methode: DNA Extraktion, PCR, NGS & Bioinformatik (5mL Lyse):
genauer: Lyse ÜN bei +56°C, DNA Extraktion über Säulen / Plattensysteme – (Unique sample tagging durch Illumina Adaptor PCR, Ligations PCR, DNA Konzentrationsmessungen an Qbit & Bioanalyzer, Miseq v2.0 2x250bp / v3.0 2x300bp – Demultiplexing, Erstellung von OTUs mit VSEARCH, OTU table, BLAST gegen Referenzdatenbanken
Vorkommen und Verteilung von Pilzarten Myzel im Waldboden Profil mittel Genanalyse
Tab. 1: Cs-137 Aktivität des Bodens und OTU reads für einige ausgewählte Pilzmyzelien. Bodenprofil Nationalpark Bayerischer Wald, Entnahmedatum: 09/2018
| reads/OTU | ||||||||||||
| Bodentiefe | Cs-137 Bq/ qm |
Elaphomyces | Boletus | Boletus | Cortinarius | Lactarius | Amanita | Amanita | Russula | Russula | Russula | |
| [cm] | spec. | edulis | spec. | olidus | lignyotus | citrina | rubescens | nigricans | ochroleuca | mustelina | ||
| 0-2 | 685 | 449 | 12 | 13 | ||||||||
| 2-4 | 2.128 | 3431 | 46 | 14 | 27 | 627 | ||||||
| 4-6 | 4.726 | 4 | 5001 | 19 | 119 | 2563 | 357 | |||||
| 6-8 | 11.553 | 1216 | 1622 | 138 | ||||||||
| 8-10 | 16.424 | 96 | 3 | 73 | ||||||||
| 10-12 | 12.859 | 35 | 2 | 6 | ||||||||
| 12-14 | 8.795 | 44 | 10797 | |||||||||
| 14-16 | 5.125 | 13632 | ||||||||||
| 16-18 | 2.891 | 15 | ||||||||||
| 18-20 | 1.600 | 20 | 26 | 36738 | ||||||||
| 20-22 | 2.309 | 33 | 19 | 17416 | 39 | 501 |
Es liegen OTU reads für viele weitere Pilz- und Bakterienarten vor. Auch weiterführende Infos zur Güte der Aussagen etc.
Die Anzahl der bestimmten OTUs ermöglichen eine grobe Abschätzung der Relationen von Myzelmengen zwischen den einzelnen Pilzarten. Exakte quantitative Aussagen für einzelne Pilzmyzele sind nicht möglich.
Ermittelte Pilzarten
Elaphomyces spec. konnten in den Bodenschichten 4-6 und vermehrt in 8-10 und 10-12 cm Bodentiefe identifiziert werden. In diesem Bodenbereich ist auch die Cs-137 Aktivität am höchsten.
Das Myzel von Russula nicricans erstreckt sich im Vergleich zu dem von Elaphomyces über das gesamt Bodenprofil und ist quantitativ dominant. Auffallend sind die hohen Zahlen an OTU reads in den tieferen Bodenschichten 18-20 und 20-22 cm.
Für Pilze werden artcharakteristische Explorationstypen angenommen. Daher sollte in einer Bodenprobe die Wahrscheinlichkeit eines Treffers für long-distance-explorationtype-Arten größer als für medium bzw. small-distance types sein. Elaphomyces ist nach allen Veröfffentlichungen (allerdings alle Agerer folgend) Short-Distance Type.
Menge und Lage der Pilzmycelien ermöglichen keine Aussagen über die Cs-137 Akkumulationsfähigkeit einzelner Pilzarten.
Ob die Ergebnisse nur mikrostandörtliche Gegebenheiten wiedergeben, oder typisch für den Waldboden in dem Gebiet sind, kann mit der Analyse von nur einem Bodenprofil nicht beantwortet werden.
Ökosysteminteraktionen zu diesem Thema sind bisher kaum erforscht. Für wissenschaftlich fundierte Aussagen werden im DNA Sequenzierungs-Bereich üblicherweise 3 Replikationen als Datengrundlage genommen.
Eine saisonale Dynamik im Jahresverlauf und in Abhängigkeit von Temperatur + Niederschlag ist zu vermuten.
Einfluss der Sampling Techniken auf die Ergebnisse sind nicht bekannt.
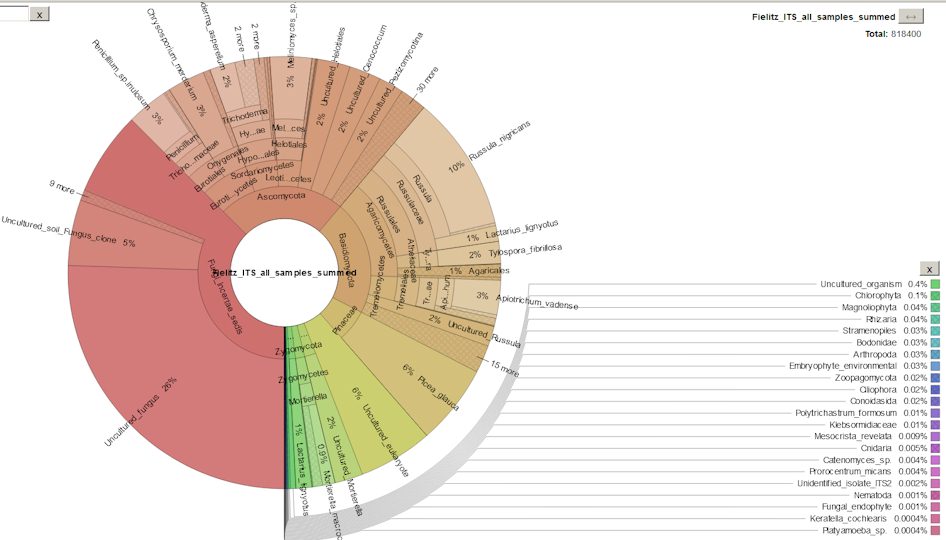
Krona Chart des gesamten Bodenprofils. Es liegen Krona Charts für jede Schichtfolge vor
Literatur
Computational Tools for Taxonomic Microbiome Profiling of Shotgun Metagenomes
OTU-Clustering im Vergleich zu exakten 16S-Sequenzvarianten
Sneath P. H. A., Sokal, R., R., 1962: Numerical Taxonomy. Nature volume 193, 855–860. Link